# 安装包
if (!requireNamespace("scRNAtoolVis", quietly = TRUE)) {
install_github('junjunlab/scRNAtoolVis')
}
if (!requireNamespace("corrplot", quietly = TRUE)) {
install.packages("corrplot")
}
# 加载包
library(scRNAtoolVis)
library(corrplot)多组火山图
多组火山图是一种用于高通量数据(如转录组、蛋白组)差异表达分析的图形。相比于传统火山图,多组火山图可以同时展示多个分组的结果,便于横向比较差异特征的一致性或特异性。
示例
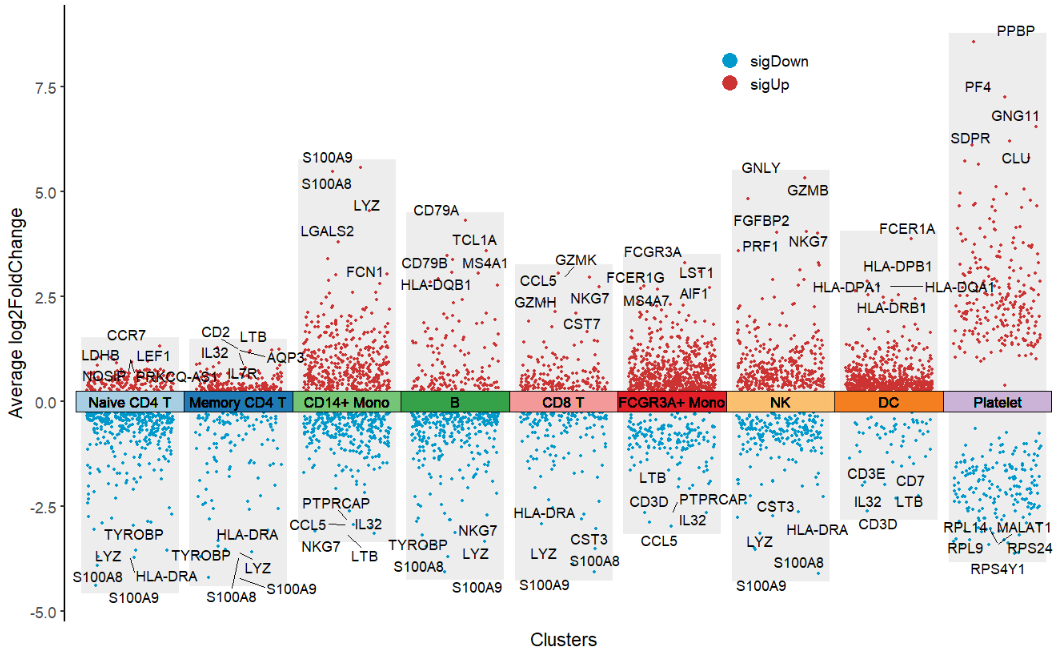
这个多组火山图展示了单细胞测序数据中多个细胞类群的差异基因表达模式。X 轴不同的颜色的方块表示不同的细胞类群,方块内部的散点表示差异基因,使用 geom_jitter 函数给散点添加了抖动效果,以避免数据点重叠。Y 轴表示差异倍数(Average log2FoldChange,红色散点表示 log2FC>0 的上调基因,蓝色散点表示 log2FC<0 的下调基因,文字标记出了 top5 差异基因。多组火山图不仅突出了各细胞类群特异性差异基因(如特定细胞类型的标记基因),还能直观识别跨细胞类群共有的关键调控分子。
环境配置
系统要求: 跨平台(Linux/MacOS/Windows)
编程语言:R
依赖包:
scRNAtoolVis;corrplot
数据准备
数据使用 scRNAtoolVis 包中提供的 pbmc.markers 单细胞测序差异基因数据集。
# 读取数据
data('pbmc.markers')
# 查看数据集
head(pbmc.markers) p_val avg_log2FC pct.1 pct.2 p_val_adj cluster gene
RPS12 2.008629e-140 0.7256738 1.000 0.991 2.754633e-136 Naive CD4 T RPS12
RPS27 2.624075e-140 0.7242847 0.999 0.992 3.598656e-136 Naive CD4 T RPS27
RPS6 1.280169e-138 0.6742630 1.000 0.995 1.755623e-134 Naive CD4 T RPS6
RPL32 4.358823e-135 0.6121027 0.999 0.995 5.977689e-131 Naive CD4 T RPL32
RPS14 3.618793e-128 0.6179756 1.000 0.994 4.962812e-124 Naive CD4 T RPS14
CYBA 1.090337e-124 -1.5739355 0.661 0.914 1.495288e-120 Naive CD4 T CYBA注意:数据集中应至少包括 p_val、avg_log2FC、p_val_adj、cluster、gene 这五列,且列名称不能改变,列顺序可以改变。绘图中展示的分组方块的名称为 cluster 列内容中的名称。
可视化
1. 基础绘图
可以使用 scRNAtoolVis 包中提供的 jjVolcano 函数绘制多组火山图。
# 基础多组火山图
p <- jjVolcano(
diffData = pbmc.markers,
topGeneN = 5,
log2FC.cutoff = 0.5,
col.type = "updown",
aesCol = c('#0099CC','#CC3333'),
tile.col = corrplot::COL2('PuOr', 15)[4:12],
cluster.order = rev(unique(pbmc.markers$cluster)),
size = 3.5,
fontface = 'italic'
)
p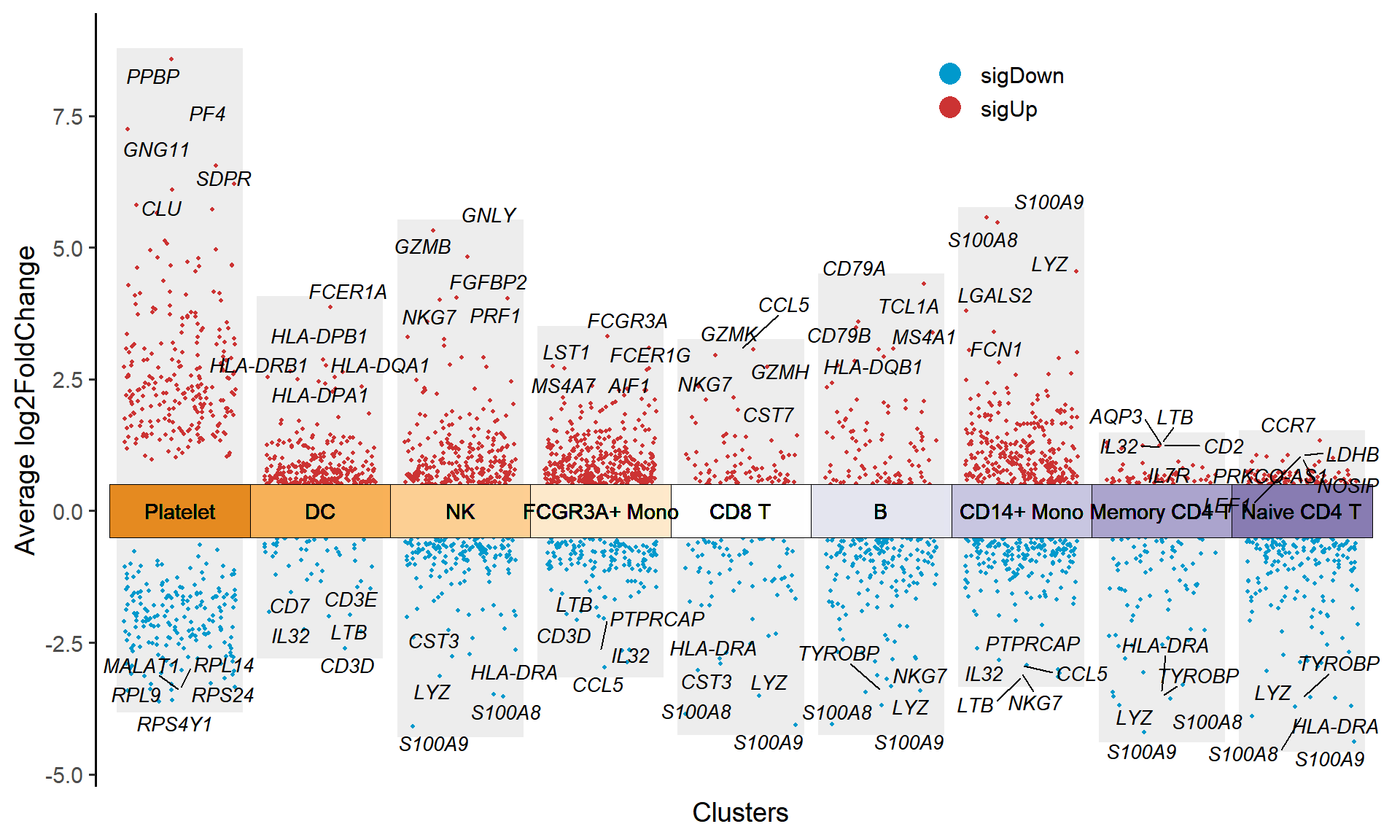
Figure 1 绘制了九种单细胞类群中上调和下调的差异基因。
关键参数:
-
diffData: 输入的差异基因表格,数据结构如pbmc.markers所示。 -
myMarkers: 自定义要展示文字标签的基因,例如:myMarkers = c('PPBP', 'PF4')自定义展示两个基因的文字标签。
# 设置参数 myMarkers = c('PPBP', 'PF4')
p <- jjVolcano(
diffData = pbmc.markers,
myMarkers = c('PPBP', 'PF4'),
log2FC.cutoff = 0.5,
col.type = "updown",
aesCol = c('#0099CC','#CC3333'),
tile.col = corrplot::COL2('PuOr', 15)[4:12],
cluster.order = rev(unique(pbmc.markers$cluster)),
size = 3.5,
fontface = 'italic',
legend.position = c(0.7,0.9)
)
p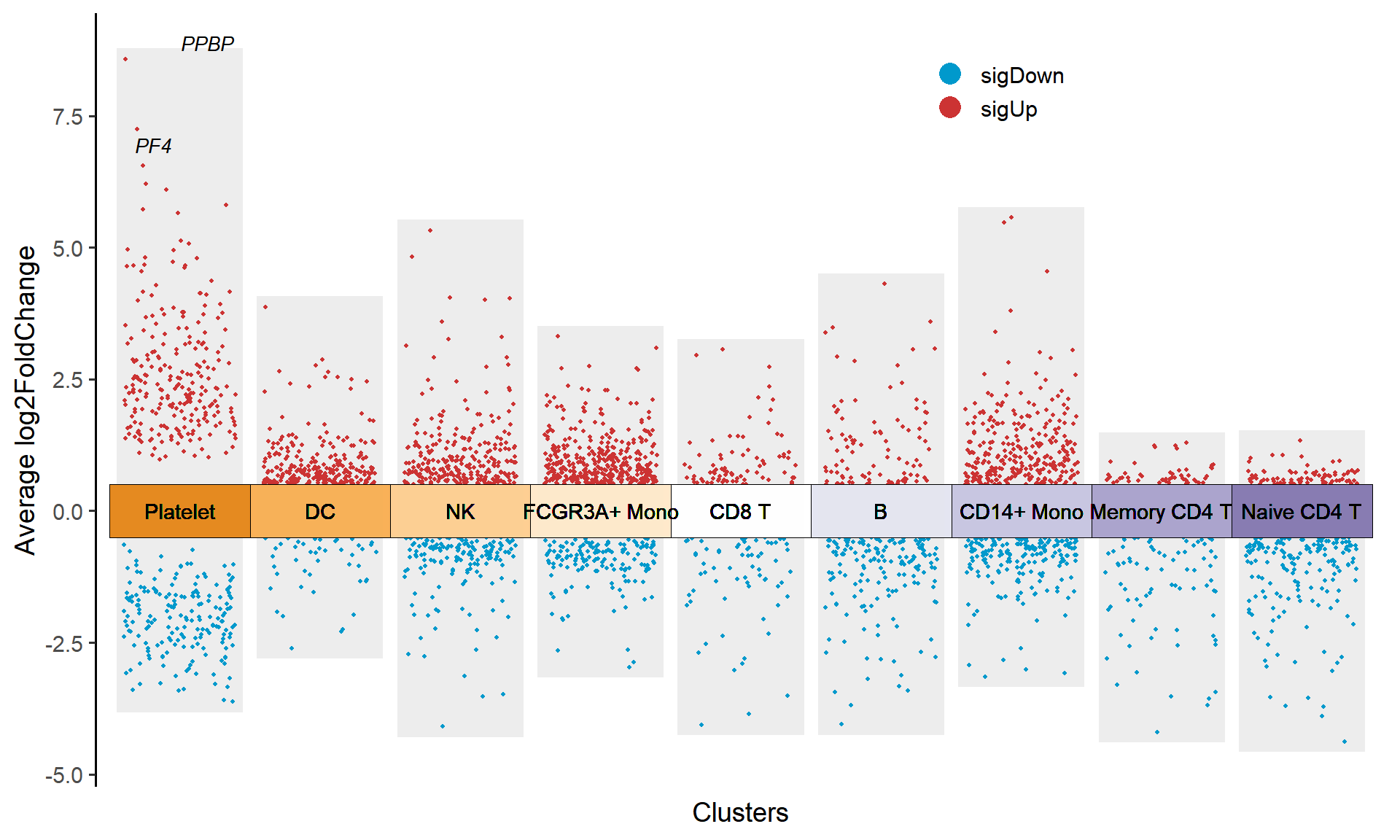
myMarkers
关键参数:
-
topGeneN: 展示多少个 top 基因的文字标签,Figure1 示例中topGeneN = 5展示了每个类群中 top5 的上调和下调基因的文字标签,topGeneN和myMarkers参数不能同时使用。 -
log2FC.cutoff: 设置 log2FoldChange 的阈值,该参数决定了分组方块的上下边界。
# 设置 log2FC.cutoff=0.5
p1 <- jjVolcano(
diffData = pbmc.markers,
topGeneN = 0,
log2FC.cutoff = 0.5,
col.type = "updown",
aesCol = c('#0099CC','#CC3333'),
tile.col = corrplot::COL2('PuOr', 15)[4:12],
cluster.order = rev(unique(pbmc.markers$cluster)),
size = 3.5,
fontface = 'italic',
legend.position = c(0.7,0.9)
)
# 设置 log2FC.cutoff=2.5
p2 <- jjVolcano(
diffData = pbmc.markers,
topGeneN = 0,
log2FC.cutoff = 2.5,
col.type = "updown",
aesCol = c('#0099CC','#CC3333'),
tile.col = corrplot::COL2('PuOr', 15)[4:12],
cluster.order = rev(unique(pbmc.markers$cluster)),
size = 3.5,
fontface = 'italic',
legend.position = c(0.7,0.9)
)
p1 + p2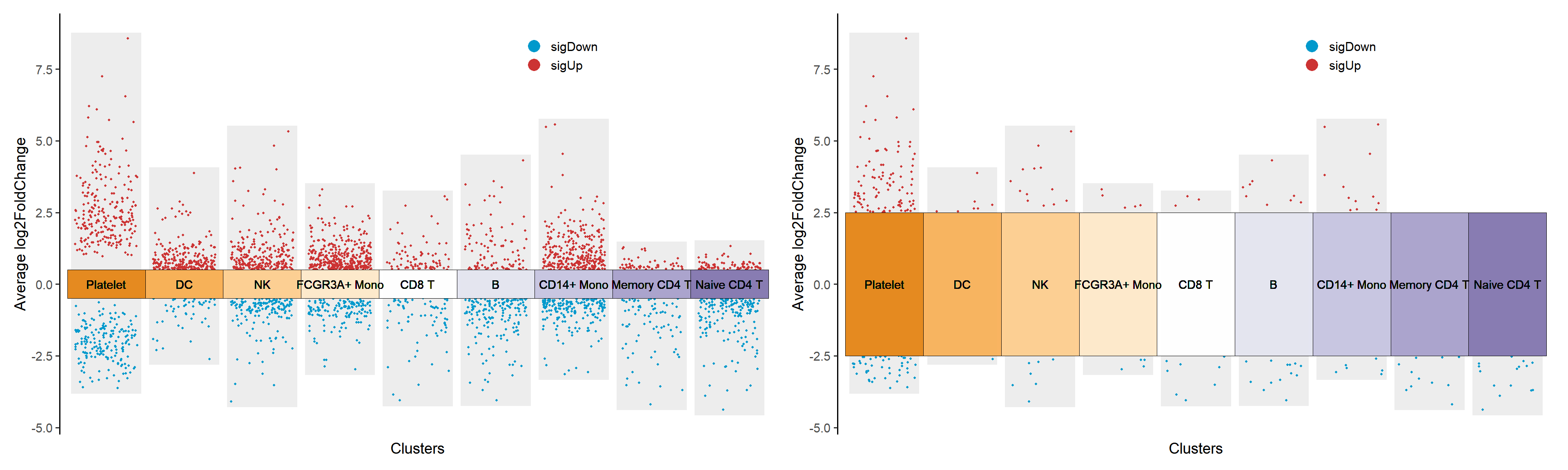
log2FC.cutoff
关键参数:
-
col.type: 散点颜色分组方式,Figure1 示例按照 “updown” 分组,也可以按照 “adjustP” 分组。
# 设置参数 col.type="adjustP"
p <- jjVolcano(
diffData = pbmc.markers,
topGeneN = 5,
log2FC.cutoff = 0.5,
col.type = "adjustP",
aesCol = c('#0099CC','#CC3333'),
tile.col = corrplot::COL2('PuOr', 15)[4:12],
cluster.order = rev(unique(pbmc.markers$cluster)),
size = 3.5,
fontface = 'italic',
legend.position = c(0.7,0.9)
)
p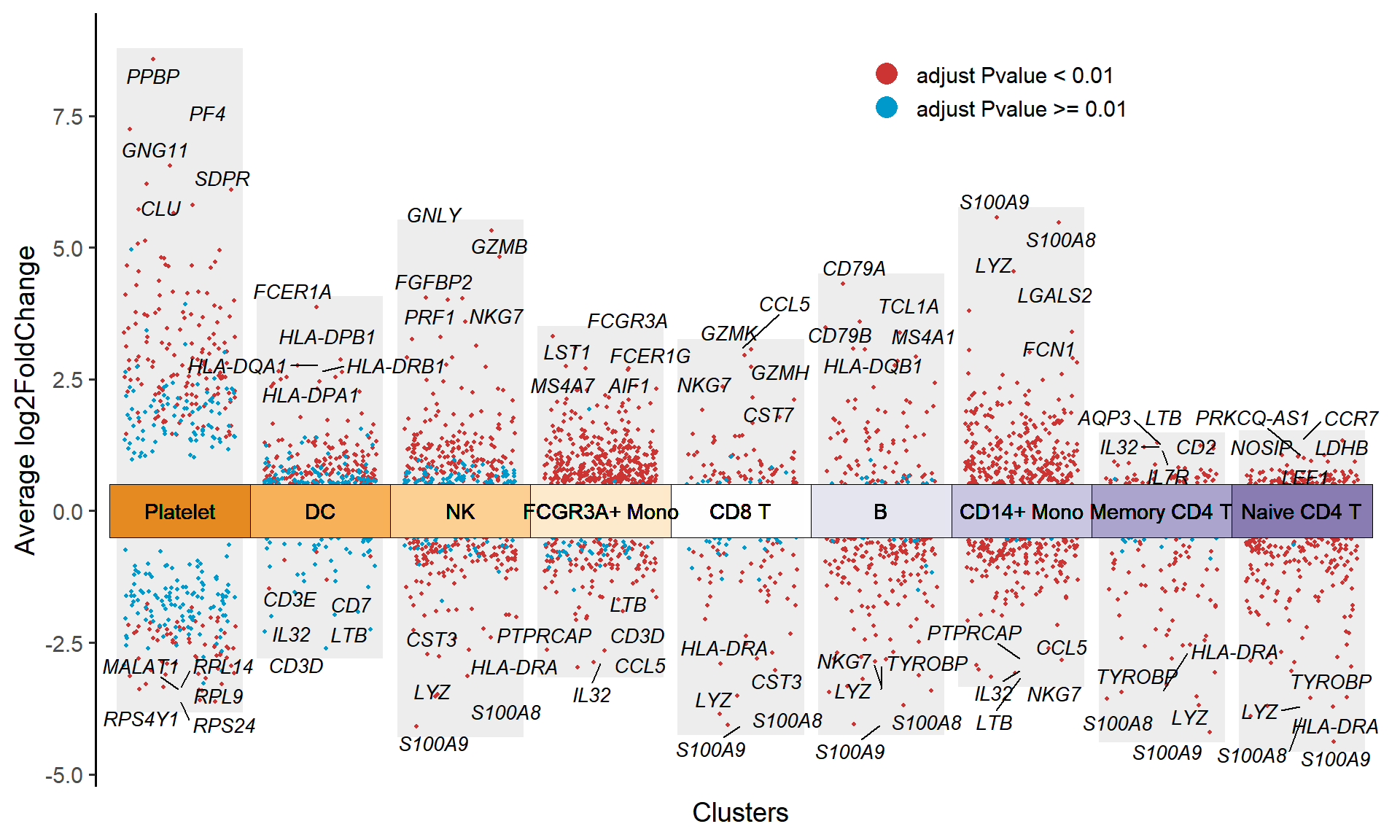
col.type
关键参数:
-
aesCol:设置散点的颜色配色。 -
tile.col:设置分组方块的颜色配色。 -
cluster.order:设置分组方块的顺序。 -
size:设置基因文字标签的字体大小。 -
fontface:设置基因文字标签的字体样式。 -
legend.position:设置 legend 的位置。
2. 旋转方向
使用 jjVolcano 函数中的 flip 参数可以旋转热图的方向。
# 横向多组火山图
p <-
jjVolcano(
diffData = pbmc.markers,
tile.col = corrplot::COL2('PiYG', 15)[4:12],
size = 3.5,
fontface = 'italic',
legend.position = c(0.8,0.2),
flip = T)
p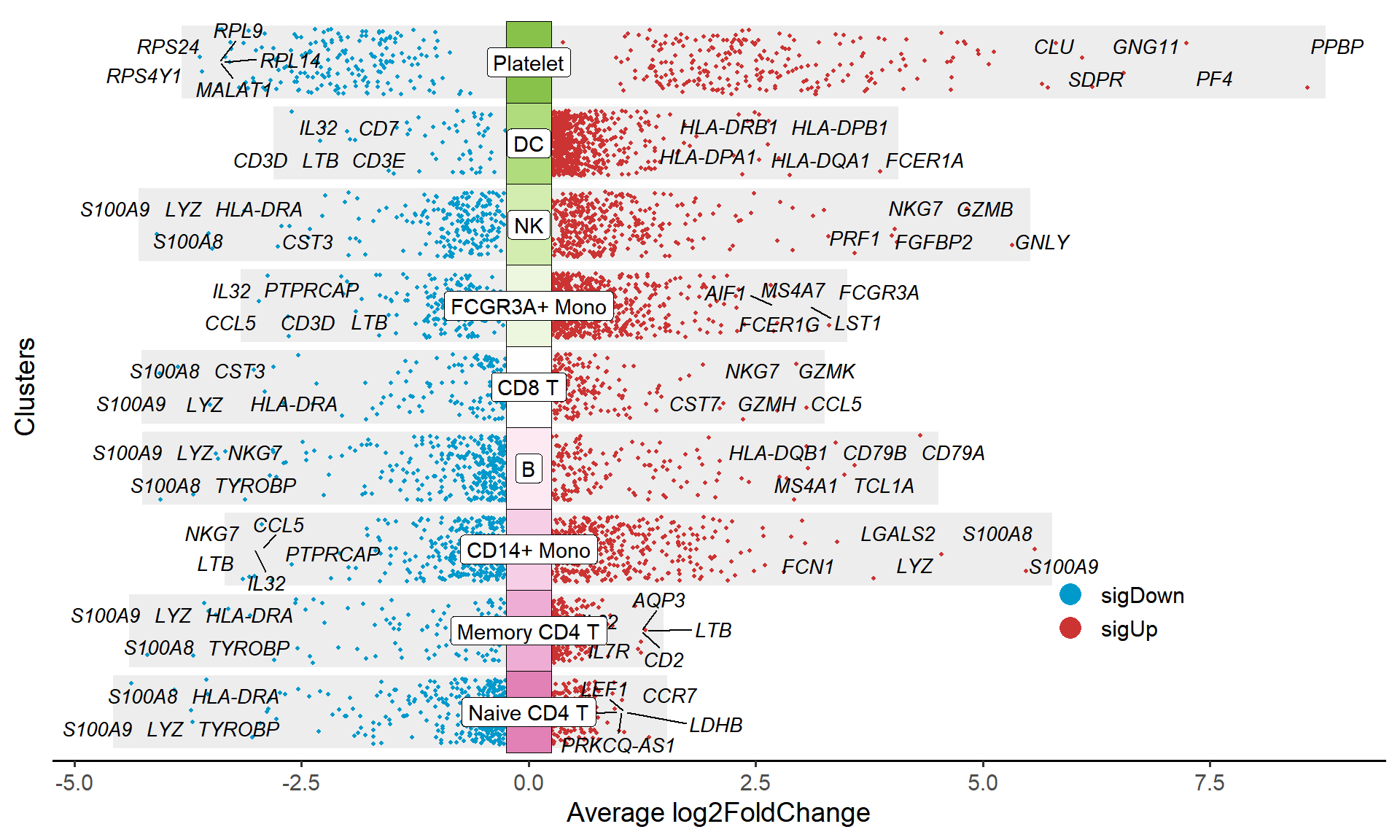
flip
Figure 5 通过设置参数 flip = T 将多组火山图旋转为横向。
3. 极坐标
# 极坐标多组火山图
p <- jjVolcano(
diffData = pbmc.markers,
tile.col = corrplot::COL2('RdBu', 15)[4:12],
size = 3.5,
fontface = 'italic',
polar = T)
p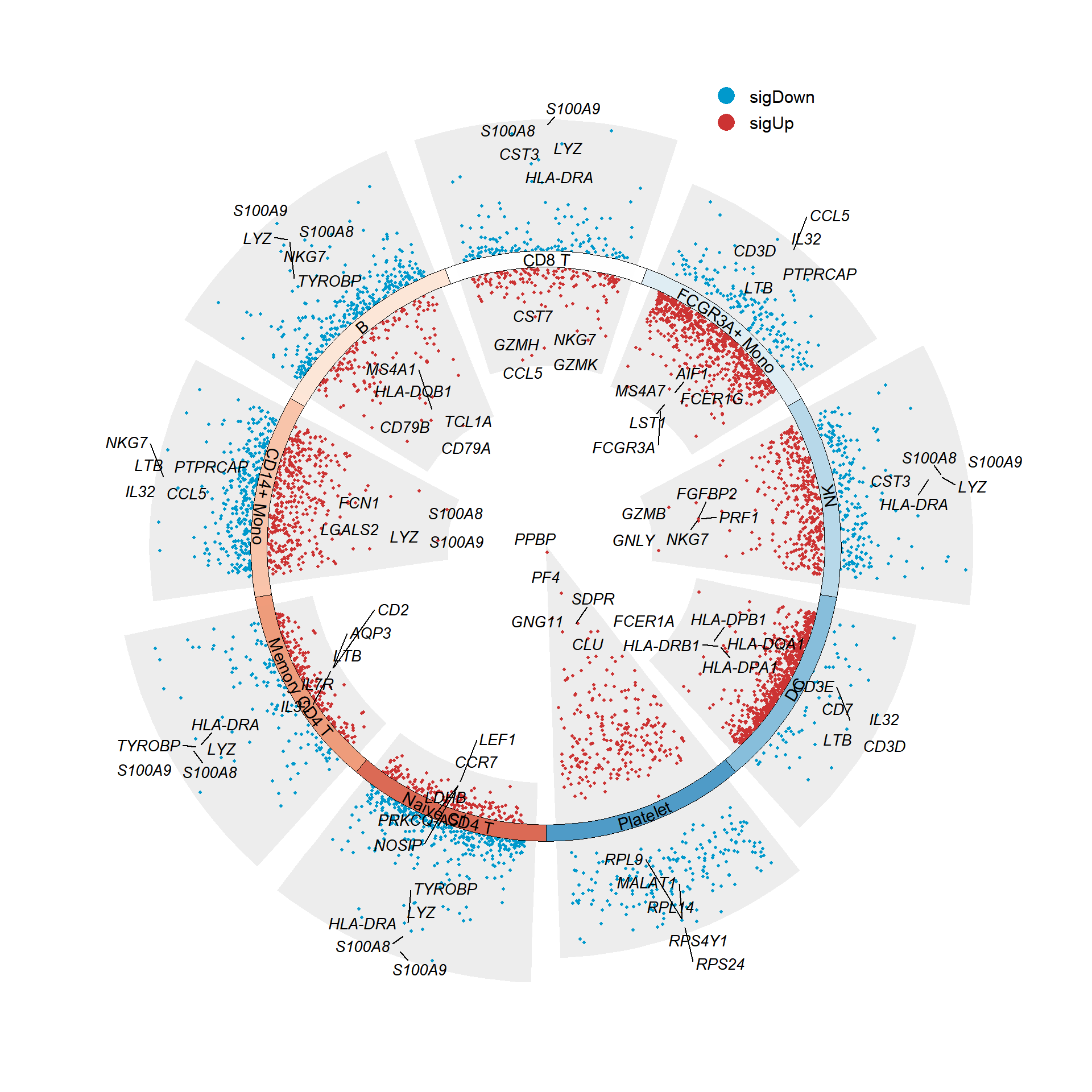
polar
Figure 6 通过设置参数 polar = T 将多组火山图转换为极坐标。
应用场景
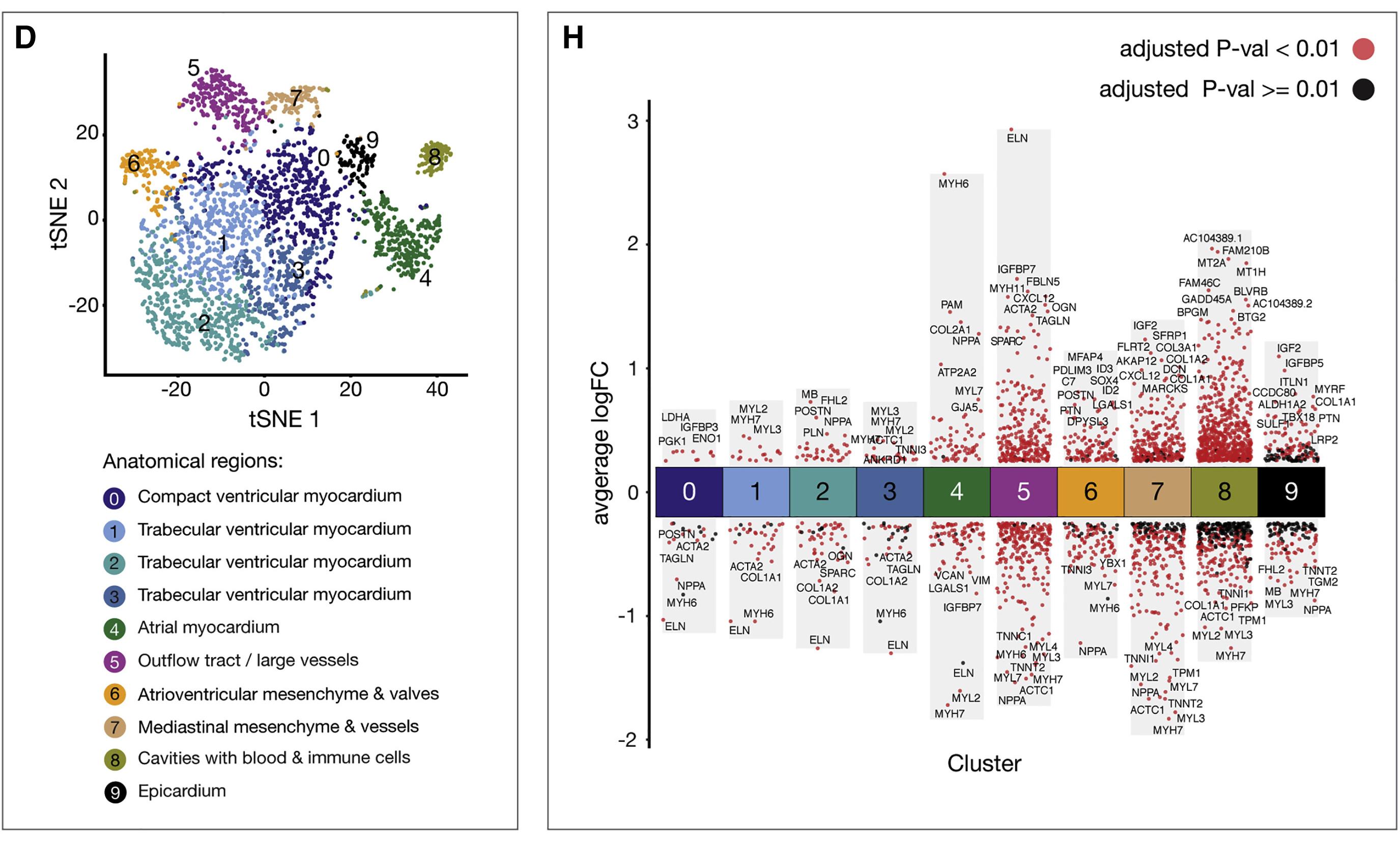
Figure 7H 显示了所有十个单细胞类群中上调和下调基因的差异基因表达分析。调整后的P值<0.01表示为红色,而调整后的P值≥0.01表示为黑色 [1]。
参考文献
[1] Asp M, Giacomello S, Larsson L, et al. A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart. Cell. 2019;179(7):1647-1660.e19. doi:10.1016/j.cell.2019.11.025.