# 安装包
if (!requireNamespace("grafify", quietly = TRUE)) {
install.packages("grafify")
}
# 加载包
library(grafify)QQ 图
注记
Hiplot 网站
本页面为 Hiplot QQ Plot 插件的源码版本教程,您也可以使用 Hiplot 网站实现无代码绘图,更多信息请查看以下链接:
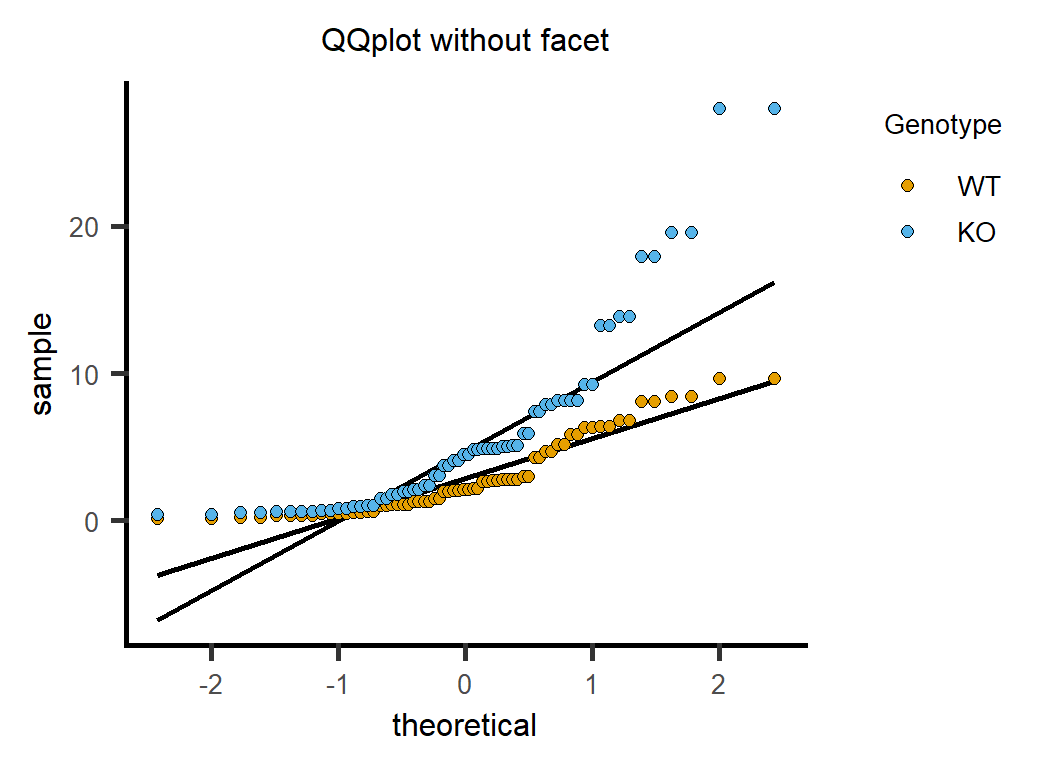
用于直观验证一组数据是否来自某个分布,或者验证某两组数据是否来自同一(族)分布。
环境配置
系统: Cross-platform (Linux/MacOS/Windows)
编程语言: R
依赖包:
grafify
数据准备
# 加载数据
data <- read.delim("files/Hiplot/148-qqplot-data.txt", header = T)
# 整理数据格式
data[, "Genotype"] <- factor(data[, "Genotype"], levels = unique(data[, "Genotype"]))
# 查看数据
head(data) Genotype Cytokine Experiment facet
1 WT 1.312322 Exp_ 1 A
2 WT 2.171466 Exp_ 2 A
3 WT 1.497610 Exp_ 3 A
4 WT 1.088861 Exp_ 4 A
5 WT 1.270528 Exp_ 5 A
6 WT 3.012708 Exp_ 6 A可视化
# QQ 图
p <- plot_qqline(data = data, ycol = Cytokine, group = Genotype,
symsize = 2, symthick = 0.5, s_alpha = 1) +
ggtitle("QQplot without facet") +
xlab("theoretical") + ylab("sample") +
guides(fill = guide_legend(title = "Genotype")) +
scale_color_manual(values = c("#E69F00","#4DB1DC")) +
theme(text = element_text(family = "Arial"),
plot.title = element_text(size = 12,hjust = 0.5),
axis.title = element_text(size = 12),
axis.text = element_text(size = 10),
axis.text.x = element_text(angle = 0, hjust = 0.5,vjust = 1),
legend.position = "right",
legend.direction = "vertical",
legend.title = element_text(size = 10),
legend.text = element_text(size = 10))
p