# 安装包
if (!requireNamespace("ggplot2", quietly = TRUE)) {
install.packages("ggplot2")
}
if (!requireNamespace("RColorBrewer", quietly = TRUE)) {
install.packages("RColorBrewer")
}
# 加载包
library(ggplot2)
library(RColorBrewer)Hi-C 热图
注记
Hiplot 网站
本页面为 Hiplot Hi-C Heatmap 插件的源码版本教程,您也可以使用 Hiplot 网站实现无代码绘图,更多信息请查看以下链接:
Hi-C 热图用于显示不同染色体上的全基因组染色质与热图的相互作用。
环境配置
系统: Cross-platform (Linux/MacOS/Windows)
编程语言: R
依赖包:
ggplot2;RColorBrewer
数据准备
加载的数据具有三个列,其中第一个用于一个基因座箱索引,第二个是另一个基因座箱索引的第二列,而这两个基因座之间的相互作用频率是第三个。
# 加载数据
data <- read.delim("files/Hiplot/087-hic-heatmap-data.txt", header = T)
# 查看数据
head(data) index_bin1 index_bin2 freq
1 135 428 13
2 365 479 38
3 209 340 8
4 216 166 34
5 288 484 5
6 162 479 14可视化
# Hi-C 热图
## 计算bins的个数
bins_num <- max(data$index_bin1) + 1
## 设置HiC数据的resolution
resolution <- 500
res <- resolution * 1000
# 设置分隔单位,以50Mb为单位进行分隔
intervals <- 50
spacing <- intervals * 1000000
## 计算breaks的数目
breaks_num <- (res * bins_num) / spacing
## 设置breaks
breaks <- c()
for (i in 0:breaks_num) {
breaks <- c(breaks, i * intervals)
}
p <- ggplot(data = data, aes(x = index_bin1 * res, y = index_bin2 * res)) +
geom_tile(aes(fill = freq)) +
scale_fill_gradientn(
colours = colorRampPalette(rev(brewer.pal(11,"RdYlBu")))(500),
limits = c(0, max(data$freq) * 1.2)
) +
scale_y_reverse() +
scale_x_continuous(breaks = breaks * 1000000, labels = paste0(breaks, "Mb")) +
scale_y_continuous(breaks = breaks * 1000000, labels = paste0(breaks, "Mb")) +
theme(panel.grid = element_blank(), axis.title = element_blank()) +
labs(title = paste0("(resolution: ", res / 1000, "Kb)"), x="", y="") +
theme_bw() +
theme(plot.title = element_text(hjust = 0.5),
legend.position = "right", legend.key.size = unit(0.8, "cm"),
panel.grid = element_blank())
p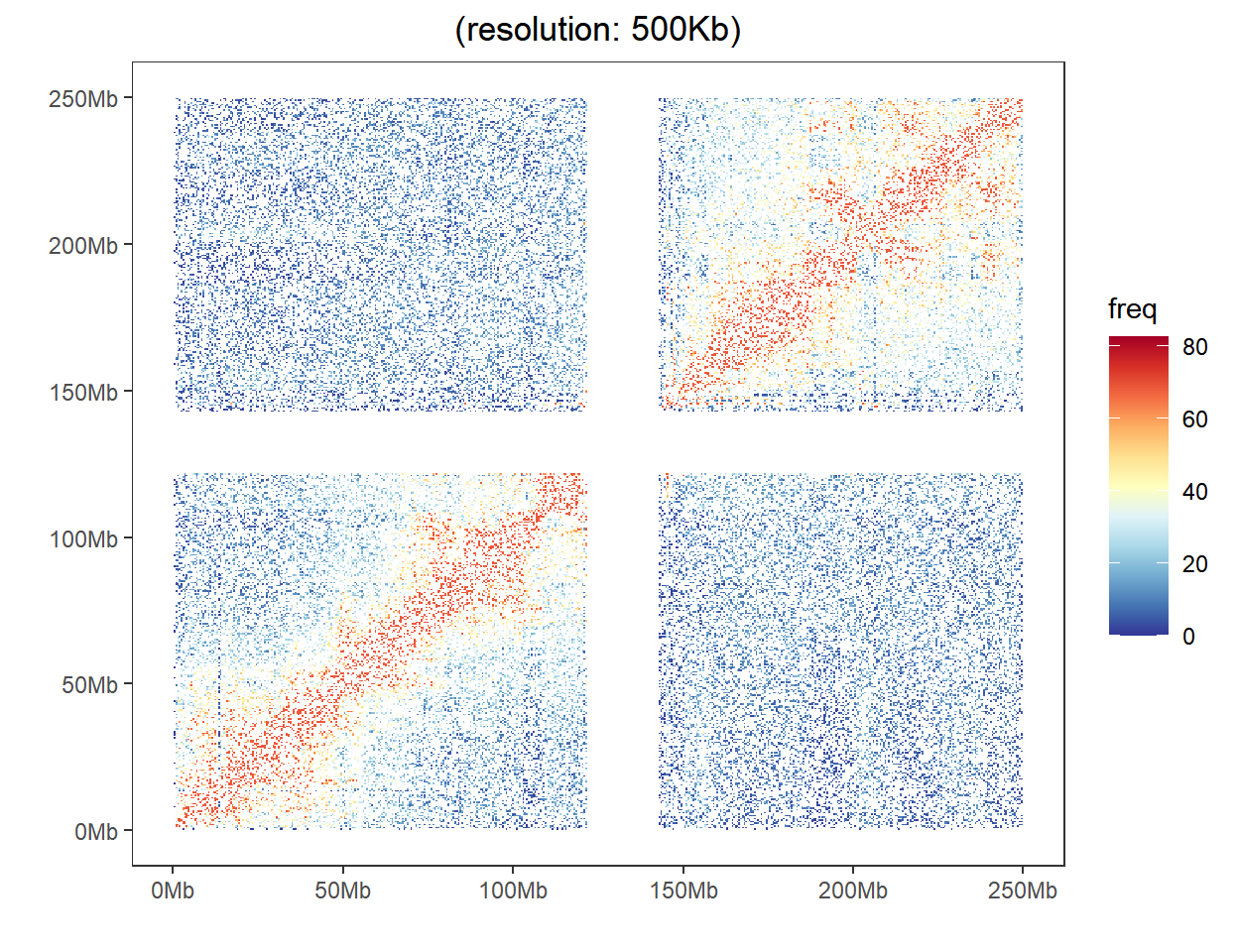
示例图每个小格表示每个基因,颜色深浅表示该基因表达量大小,表达量越大颜色越深(红色为上调,绿色为下调)。每行表示每个基因在不同样本中的表达量状况,每列表示每个样本所有基因的表达量情况。上方树形图表示不同组群和年龄的不同样本的聚类分析结果,左侧树形图表示来自不同样本的不同基因的聚类分析结果。