# 安装包
if (!requireNamespace("survminer", quietly = TRUE)) {
install.packages("survminer")
}
if (!requireNamespace("survival", quietly = TRUE)) {
install.packages("survival")
}
if (!requireNamespace("ggplotify", quietly = TRUE)) {
install.packages("ggplotify")
}
# 加载包
library(survminer)
library(survival)
library(ggplotify)生存分析
注记
Hiplot 网站
本页面为 Hiplot Survival Analysis 插件的源码版本教程,您也可以使用 Hiplot 网站实现无代码绘图,更多信息请查看以下链接:
生存曲线是显示特定物种或群体 (如雄性或雌性) 在每个年龄存活下来的个体数量或比例的图表。
环境配置
系统: Cross-platform (Linux/MacOS/Windows)
编程语言: R
依赖包:
survminer;survival;ggplotify
数据准备
载入数据为时间点,生存状态及分组。
# 加载数据
data <- read.delim("files/Hiplot/169-survival-data.txt", header = T)
# 整理数据格式
colnames(data) <- c("Time", "Status", "Group")
data[,1] <- as.numeric(data[,1])
fit <- survfit(Surv(Time, Status == 1) ~ Group, data = data)
data <- data[data[,1] < 1100,]
# 查看数据
head(data) Time Status Group
1 306 1 G1
2 455 1 G1
3 1010 0 G1
4 210 1 G1
5 883 1 G1
6 1022 0 G1可视化
# 生存分析
p <- ggsurvplot(
fit, data = data, risk.table = T, pval = T, conf.int = T, fun = "pct",
size = 0.5, xlab = "Time", ylab = "Survival probability",
ggtheme = theme_bw(), risk.table.y.text.col = TRUE,
risk.table.height = 0.25, risk.table.y.text = T,
ncensor.plot = T, ncensor.plot.height = 0.25,
conf.int.style = "ribbon", surv.median.line = "hv",
palette = c("#00468BFF", "#ED0000FF"),
xlim = c(0, 1100), ylim = c(0, 100),
break.x.by = 150)
p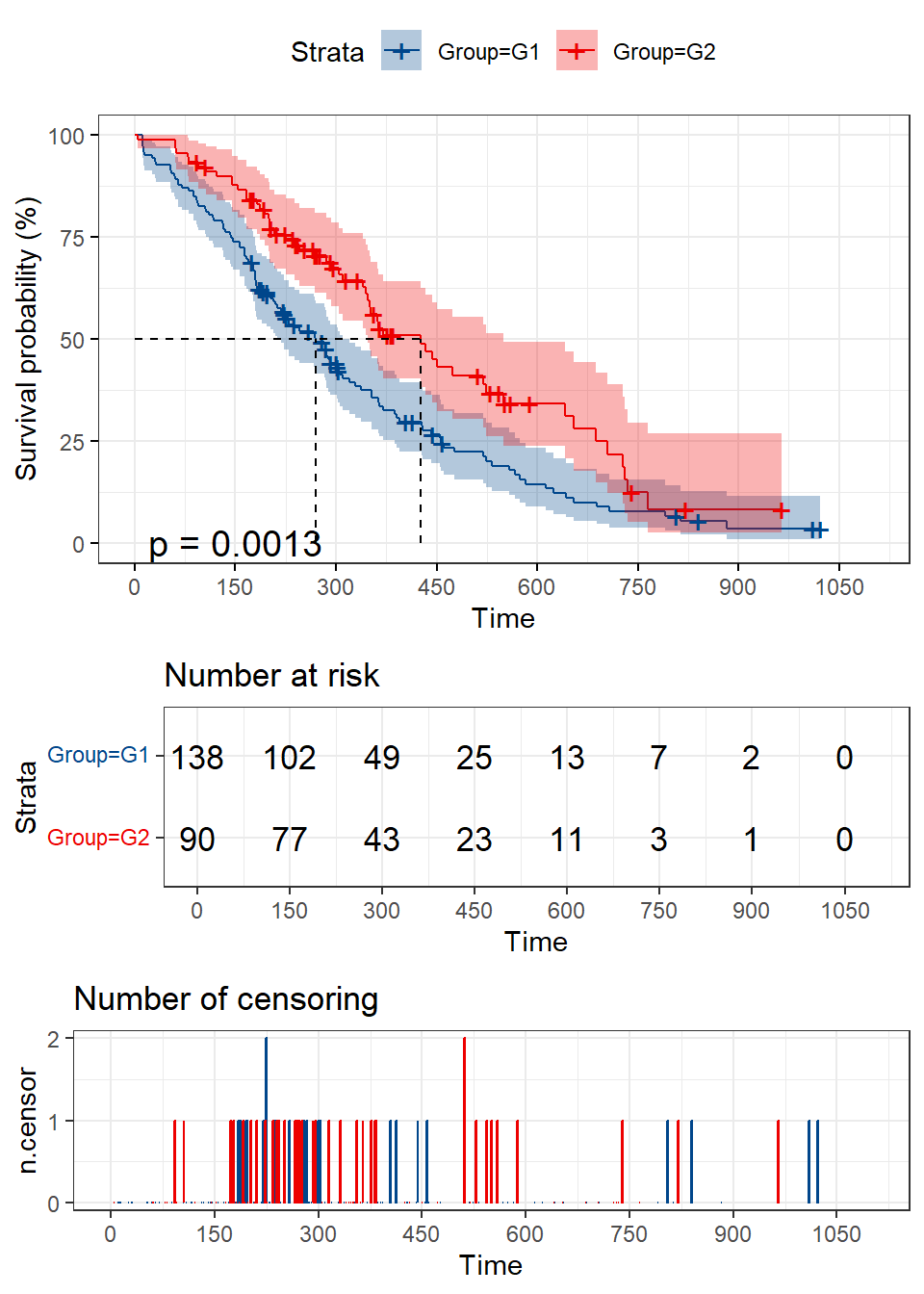
横轴表示时间轴,纵轴表示生存概率。蓝色曲线表示 G1 组生存曲线,红色曲线表示 G2 组生存曲线。经过 logrank 检验后发现 P 值 = 0.0013 < 0.05,表明两组患者生存状况的差异不能用抽样误差来解释,分组因素才是导致两条曲线生存率出现差异的原因。该示例图形表明,G2 组的总体生存率要好于 G1 组。